* 코로나19 검사 방법
의심 환자의 침이나 가래 등 가검물 채취 => 핵산(RNA) 추출 => PCR 검사
* 핵산 추출 방법(Nucleic acid extraction methods)
출처 : https://m.blog.naver.com/sml-meditree/221722169513 발췌 및 수정
1. 페놀 추출법
세포를 포함하는 시료를 SDS나 프로테이나아제 (proteinase) K로 처리하여 가용화한 후 페놀로 단백질을 변성 제거하여 핵산을 정제하는 방법.
=> 페놀 추출법은 여러 단계를 포함하여 시간이 많이 소요되며, 핵산 추출 효율은 작업자의 노련성에 따라 크게 좌우된다.
2. 컬럼을 이용한 키트(핵산 추출의 기본)
핵산과 특이적으로 결합하는 실리카나 유리 섬유 필터를 이용한 방법으로, 세포를 카오트로픽 시약(chaotropic reagent)으로 처리하여 세포를 용해시키고, 물분자와 핵산 사이의 구조적 상호작용의 원리를 이용하여 핵산 분자를 단백질 및 기타 세포내 물질로부터 정제하는 방법이다.
유리섬유나 실리카 막은 단백질, 세포 대사 물질들과 결합 비율이 낮으므로 상대적으로 높은 농도의 핵산을 얻을 수 있다. 이 방법은 페놀법과 비교하면 간편하지만, PCR등의 효소 반응을 강하게 저해 시키는 카오트로픽 시약이나 에탄올을 이용하기 때문에 이들 물질을 완전히 제거(세정)해야 하며, 조작이 복잡하고 많은 시간이 걸리는 단점이 있다.
3. 핵산을 직접 정제하는 방법(국제 공개 WO 00/21973호)
시료를 필터에 통과시켜 세포를 필터에 흡착시킨 후 필터에 흡착된 세포를 용해시키고 필터로 여과시킨 후 필터에 흡착된 핵산을 세정, 용출 시킨다.
세포를 필터에 흡착시킨 후 핵산을 용출 시키기 위해서는 세포의 종류에 따라 필터를 선택하여야 한다.
- Solution-based methods
- Solid-phase based methods
. DNA Extraction
_출처 : https://ko.wikipedia.org/wiki/DNA_%EB%B6%84%EB%A6%AC_
DNA 분리(DNA extraction)는 1869년 프리드리히 미셔(Friedrich Miescher)에 의해 만들어졌다.[1] 현재 이것은 분자생물학이나 법의학에서 자주 사용되는 기법이다. 화학적 방법의 경우 추출에 사용되는 다양한 키트가 있으며 본인의 목적에 맞는 올바른 키트를 선택해야 추출 절차에 소요되는 시간을 절약할 수 있다.
● 기본 절차
1) 연구할 세포를 수집해야 한다. 이때 수집의 양은 사용하는 방법에 따라 다르다.
2) 세포막을 부수면 내부의 세포질과 함께 DNA가 노출된다(세포 용해).
o 세포막과 핵의 지질은 세제와 계면활성제로 분해된다.[a]
3) 이 용액을 농축된 염 용액(식염수)으로 처리하여 부서진 단백질, 지질 및 RNA와 같은 파편을 함께 덩어리로 만든다.
4) 덩어리진 세포 파편을 DNA에서 분리하기 위해 용액을 원심 분리한다.
5) 세포 용해 단계에서 사용되는 세제, 단백질, 염 및 시약으로부터 DNA를 정제한다. 이때 가장 일반적으로 사용되는 절차는 다음 3가지이다.
o 일반적으로 0°C에 가까운 에탄올 또는 이소프로판올에 의한 에탄올 침전. DNA는 이러한 알코올에 녹지 않기 때문에 함께 응집되어 원심분리 시 펠릿을 생성한다. 일반적으로 아세트산 나트륨을 첨가하여 이온 강도를 증가시켜 DNA의 침전을 개선한다.
o 페놀로 하여금 시료의 단백질을 변성시키게 하는 페놀-클로로포름 추출. 샘플을 원심분리한 후 변성된 단백질은 유기상에 머무르고 핵산을 함유한 수상은 클로로포름과 혼합되어 용액에서 페놀 잔류물을 제거한다.
o 버퍼의 pH 및 염 농도에 따라 핵산이 고체상(실리카 또는 기타)에 결합(흡착)할 수 있다는 사실에 의존하는 미니컬럼 정제.
DNA에 결합된 세포 및 히스톤 단백질은 프로테아제를 첨가하거나, 나트륨 또는 암모늄 아세테이트로 단백질을 침전시키거나, DNA 침전 전에 페놀-클로로포름 혼합물로 추출하는 방법 등으로 제거할 수 있다.
분리 후, DNA는 일반적으로 TE 완충용액 또는 초순수에 있는 약알칼리성 완충용액에 용해된다.
● DNA 분리 방법
가장 일반적인 DNA 추출 방법에는 유기 추출, Chelex 추출 및 고체상 추출이 있다.[3] 이러한 방법 모두 분리된 DNA를 산출하지만, 산출되는 DNA의 품질, 양 등은 전부 다르다. 그래서 DNA 추출 방법을 선택할 때는 비용, 시간, 안전성, 오염 위험 등 여러 요소를 고려해야 한다. 다양한 생물학적 샘플에 대해 상업적으로 이용 가능한 DNA 추출 키트가 있다.
1) 유기 추출
유기 추출에는 여러 다른 화학 용액을 추가하고 배양하는 것이 포함된다.[3] 용해 단계, 페놀(중성) 클로로포름 추출, 에탄올 침전 및 세척 단계를 포함한다. 유기 추출은 값이 싸고 순수한 DNA를 대량으로 생성하기 때문에 실험실에서 자주 사용된다. 쉽지만 여러 단계가 필요하고 다른 방법보다 시간이 오래 걸린다. 또한 독성 화학물질인 페놀과 클로로포름의 바람직하지 않은 사용과 관련이 있으며 여러 튜브 사이에 DNA를 전달하여 오염 위험이 증가한다.[4] DNA의 유기적 추출을 기반으로 하는 여러 프로토콜이 수십 년 전에 효과적으로 개발되었지만[5] 이러한 프로토콜의 개선되고 실용적인 버전도 지난 몇 년 동안 개발 및 출판되었다.[6]
2) Chelex 추출
Chelex 추출 방법은 Chelex 수지를 시료에 첨가하고 용액을 끓인 다음 와동 및 원심분리하는 것이다. 세포 물질은 Chelex 구슬에 결합하는 반면 DNA는 상등액에서 사용할 수 있다.[4] Chelex 방법은 유기 추출보다 훨씬 빠르고 간단하며 하나의 튜브만 필요하므로 DNA 오염 위험이 줄어든다. 그러나 Chelex 추출도 단점이 있다. 많은 양을 산출하지 못하며, 산출된 DNA는 단일 가닥이므로 RFLP가 아닌 PCR 기반 분석에만 사용할 수 있다.[4]
3) 고체상 추출
스핀 컬럼 기반 추출 방법을 사용하는 것과 같은 고체상 추출은 DNA가 실리카에 결합한다는 사실을 이용한다. DNA를 포함하는 샘플은 실리카 겔 또는 실리카 비드 및 카오트로픽 염을 포함하는 컬럼에 추가된다. 카오트로픽 염은 가닥 사이의 수소 결합을 방해하고 핵산을 소수성으로 만들어 DNA와 실리카의 결합을 촉진한다. 이것은 인산염 잔류물을 노출시켜 흡착에 사용할 수 있다.[7] DNA는 실리카에 결합하고 나머지 용액은 에탄올을 사용하여 씻어내어 카오트로픽 염 및 기타 불필요한 구성 요소를 제거한다.[3] 그런 다음 DNA는 저염 수용액으로 재수화되어 비드에서 DNA가 용출될 수 있다.
이 방법은 PCR 및 RFLP 분석 모두에 사용할 수 있는 고품질의 대부분 이중 가닥 DNA를 생성한다. 이 절차는 자동화할 수 있으며[4] 페놀-클로로포름 방법 보다 낮지만 처리량이 높다. 이것은 1단계 방법이다. 즉, 전체 절차가 하나의 튜브에서 완료된다. 이것은 오염 위험을 낮추어 DNA의 법의학 추출에 매우 유용한다. 여러 고체상 추출 상용 키트는 여러 회사에서 제조 및 판매한다. 유일한 문제는 유기 추출 또는 Chelex 추출보다 비용이 많이 든다는 것이다.
● 특수 유형의 DNA 분리 방법
일부 샘플에서 DNA를 분리하려면 특정 기술을 선택해야 한다. 복잡한 DNA 분리가 있는 일반적인 샘플은 다음과 같다.
· 부분적으로 분해된 DNA를 포함하는 고고학 샘플.[8]
· 토양, 남색 및 기타 직물 염료의 부식산 또는 혈액 내 헤모글로빈과 같은 후속 분석 절차의 억제제, 특히 PCR 억제제를 포함하는 샘플.
· 효모와 같은 두꺼운 세포벽을 가진 미생물의 샘플.
· 여러 출처의 혼합 DNA를 포함하는 샘플.
Extrachromosomal DNA는 일반적으로 분리하기 쉽다. 특히 플라스미드는 세포 용해 후 단백질 침전에 의해 쉽게 분리될 수 있으며, 이는 염색체 DNA를 불용성 분획에 가두고 원심분리 후 플라스미드 DNA를 가용성 분획으로부터 정제할 수 있다.
Hirt DNA 추출은 포유동물 세포에서 모든 염색체 외 DNA를 분리하는 것이다. Hirt 추출 과정은 고분자량 핵 DNA를 제거하고 세포에 존재하는 저분자량 미토콘드리아 DNA와 바이러스 에피솜만 남긴다.
● 고분자량 DNA 추출법
이 방법에서 식물 핵은 물리적으로 조직을 분쇄하고 고유한 핵 분리 완충용액(NIB)에서 손상되지 않은 핵을 재구성하여 분리된다. plastid DNA는 세포 소기관에서 방출되고 세척 및 원심분리에 의해 삼투 완충용액으로 제거된다. 그런 다음 정제된 핵을 용해하고 유기 추출에 의해 추가로 세척하고 genomic DNA를 고농도 CTAB로 침전시킨다. 고순도의 고분자량 gDNA를 핵에서 추출하여 높은 pH 완충용액에 녹여 안정적으로 장기간 보관할 수 있다.[9]
● DNA 검출
디페닐아민(DPA) 지시약은 DNA의 존재를 확인할 것이다. 이 절차는 DNA의 화학적 가수분해를 포함한다. 가열될 때(예: ≥95°C) 산에서 반응은 데옥시리보스 당을 필요로 하므로 DNA에 특이적이다. 이러한 조건에서 2-데옥시리보스는 w-히드록시레불리닐 알데히드로 전환되고, 이는 화합물인 디페닐아민과 반응하여 청색 화합물을 생성한다. DNA 농도는 600nm에서 용액의 흡광도 강도를 측정하여 결정할 수 있다. 분광광도계로 흡광도를 측정하고 알려진 DNA 농도의 표준 곡선과 비교한다.
260nm 및 280nm 파장에서 DNA 용액의 흡광도를 측정하는 것은 DNA 순도의 척도로 사용된다. DNA는 제한 효소로 DNA를 절단하고, 아가로스 겔에서 이를 실행하고, 브로민화 에티듐(EtBr) 또는 다른 염색제로 염색하고, DNA의 강도를 알려진 농도의 DNA 마커와 비교하여 정량할 수 있다.
서던 블롯 기술을 사용하여 이 정량화된 DNA를 분리하고 PCR 및 RFLP 분석을 사용하여 추가로 검사할 수 있다. 이러한 절차는 게놈 내에서 반복되는 서열의 분화를 허용한다. 법의학 과학자들이 비교, 식별 및 분석에 사용하는 것은 바로 이러한 기술이다.
. RNA Extraction Process
RNA는 단일나선(single strand) 구조를 가져 세포내 RNases(1) 및 다양한 외부 자극에 의하여 분해되거나 원형 보존이 어렵다. 현재까지 개발되어진 RNA 분리 방법들은 RNA 손실을 최소화하고 세포, 조직 등의 샘플로부터 고효(수)율, 고순도로 분리될 수 있도록 개발되었으며 상대적으로 저렴한 수동방법(Manual method)부터 kit를 사용하여 분리하는 등의 다양한 방법들이 있다. (1) RNA를 분해하는 효소
● RNA 분리 방법
1) Acid phenol extraction and alcohol precipitation (Phenol ethanol precipitation)
핵산은 극성이 높아 phenol 존재시에 용해되지 않고 밀도도 물보다 높기 때문에 phenol이 sample에 첨가되면 수성층(aqueous phase) 및 유기층(organic phase)을 형성하며 분리된다. 이때 수성층에 존재하는 RNA를 LiCl(Lithium Chloride)와 에탄올(EtOH)를 이용 침전 분리한다.
- 단점: 분리 과정이 길고 복잡, Sample에 phenol이 잔존할 수 있음, RNases를 즉시 불활성화 시킬 수 없음.
- 장점: 가격이 저렴함.
2) TRizol or tri followed by chloroform and precipitation
TRizol을 구성하는 여러 성분 중 guanidine isothiocyanate은 강력한 RNase 억제제로 phenol(Trizol이나 DW에 saturaton시킨 알카리성)은 세포내의 단백질과 지질을 붕괴시킨다. 붕괴되어진 혼합물에 chloroform을 첨가하면 수성층과 유기층으로 분리되어 수성층에 존재하는 RNA를 Isopropanol로 침전 분리하는 방법으로, 침전되어진 RNA는 TE buffer 또는 RNase free water로 용해시켜 얻는다.
- 단점: 유독성 유해물질(phenol, chloroform, isopropanol) 사용, DNA 오염 가능성 있음.
- 장점: 추출 과정 중 RNA가 변성없이 보호되며, manual 방법 중 RNA의 회수율과 순도가 높음.

. Trizol(Tri-reagent)을 이용한 RNA 분리
참고: https://www.thermofisher.com/order/catalog/product/10296028?SID=srch-hj-10296-028
Matrices표면에 양전하를 띤 DEAE(Diethylaminoethyl cellulose)와 음전하를 띤 RNA의 상호 작용에 기초하여 분리하는 방법으로 Anion-exchange matrices는 Silica bead로 구성되며, 이는 넓은 구멍과 높은 전하 밀도를 갖는 친수성(hydrophilic)표면으로 코팅되어 있다. Buffer의 pH조건과 염분 농도에 따라 DNA와 RNA가 용출되는 방법이다.
- 특징: Genomic DNA와 RNA 동시에 정제 가능
양전하를 띠는 Silica 입자와 음전하를 띠는 RNA분자의 높은 친화도를 이용하여 높은 염분 조건에서 RNA가 matrices에 단단하게 결합하는 원리를 사용하는 방법으로, DNase를 처리 잔류 DNA를 제거한 후 RNA는 TE buffer 또는 distilled water를 이용하여 용출한다.
- 장점: RNA 분리에 널리 사용되는 방법으로 빠른 추출과 높은 수율 가능
- 단점: 고가이며, 200nt 이하의 짧은 정제에는 적합하지 않음(miRNA를 얻는 Norgen column제외)
5) Commercially available kits
Spin Column 내부에 anion-exchange column 또는 silica column등의 원리를 이용하는 column을 사용하여 분리하는 방법으로, 덮개가 있어 sample의 RNase free 상태를 보장하여 전체적인 오염을 방지할 수 있으며, 개별 포장으로 1개의 sample을 각각으로 사용할 수 있다. Phenol과 같은 유해 물질이 kit에 포함되지 않기 때문에 공기 정화 후드를 반드시 필요로 하지 않는다. (예: Qiagen RNeasy kit-silica column사용 고순도, 고수율 DNA추출)
- 장점: 소요 시간이 짧고, 유해 화학물질이 없으며, column의 손상이 없으면 RNA의 손상 없음.
- 단점: 200nt보다 작은 RNA는 제거되므로 최대 산출량이 낮고 manual방법에 비하여 가격이 높음.
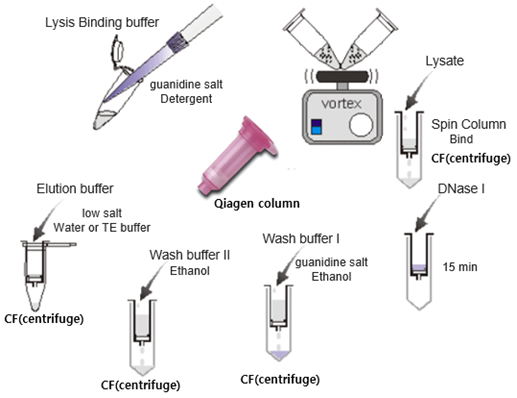
. RNA purification using spin column
● RNA 분리의 목적 및 활용
RNA는 세포 분열, 분화 및 성장과 세포 노화, 사망 등에 이르기까지 세포의 전 생애 조절에 중요한 역할을 하며, 특정 RNA의 결함이나 조절 작용은 심장병, 일부 암, 뇌졸중 등을 포함하는 많은 질병과도 연관이 있다. RNA는 DNA와 달리 세포의 유형, 외부 자극 또는 질병의 유무에 따라 유전자 발현 패턴 및 RNA분해 정도가 다르게 나타나기 때문에 다양한 연구에 활용될 수 있어 그 이용 가치가 높다. 또한 바이러스를 매개로 하는 임상 질병의 대부분은 RNA를 통하여 유전 정보를 운반하므로 관련 분야 연구에 RNA분리는 매우 중요하게 활용될 수 있다.
이전 RNA interference(RNAi)를 이용한 Gene silencing 연구로부터 최근의 DNA-sequencing 및 RNA-sequencing 연구가 유전자 발현 연구에 핵심 역할을 하고 있으며, 다양한 DNA-sequencing 분석을 통한 유전자 탐색과 발현 조절 기작(생물의 생리적인 작용을 일으키는 기본 원리) 탐색, 개별 유전자의 상세 판독 등과 더불어 환경에 따라 상대적으로 다르게 발현되는 RNA를 발견하고 그 발현 값을 정량화 하는데 이용되는 등 RNA분석은 기초분야는 물론 응용분야에서도 주목을 받고 있다.
핵산 추출 장치의 예

Solution-based method
Many of the early methods for nucleic acid extraction depended on grinding frozen biological samples, and then mixing them with solutions of chemicals devised to make it possible to purify RNA and/or DNA.
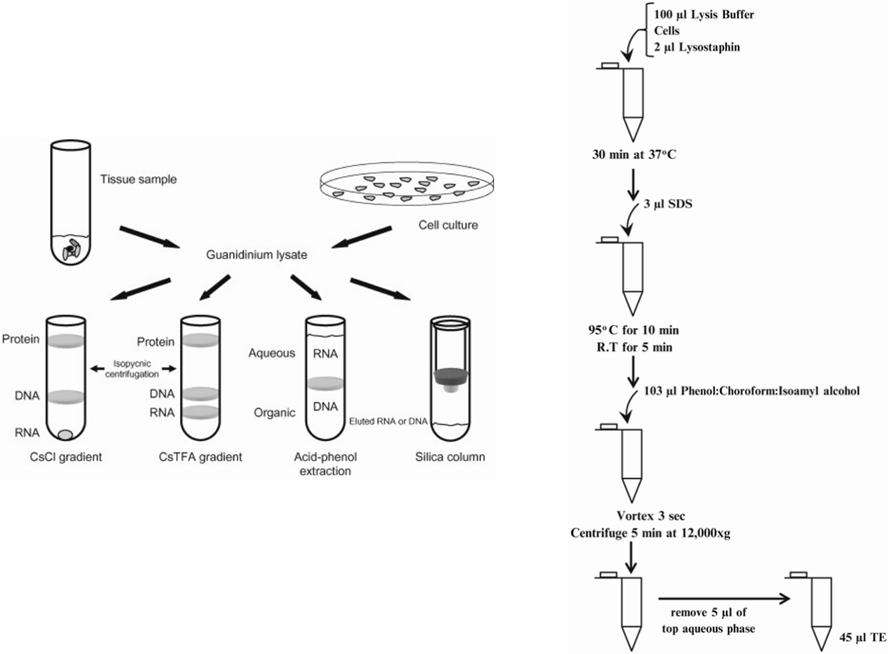
1. Cesium Chloride Gradient Centrifugation (with Ethidium Bromide)
This method is based on the phenomena of buoyant and specific density. Cesium chloride (CsCl) is an extremely dense salt. When solutions of this salt are subjected to centrifugation at very high speeds (typically >100,000 rpm), the CsCl sets up a concentration gradient, where it is highly concentrated at one end, or side, of the centrifuge tube that it’s in, and much less concentrated at the other. If the centrifugation process is ended gently, with the centrifuge slowing down slowly, the denser solution (with a higher CsCl concentration) will settle to the bottom of the tube, maintaining the concentration gradient for some time. Other molecules dissolved in the solution will separate themselves according to how dense they are, with the denser molecules going toward the bottom of the tube, and less dense molecules moving toward the top. When DNA is in the presence of a molecule called ethidium bromide, it will bind to it and become fluorescent when under UV light. The ethidium bromide also adjusts the density of DNA so that it moves toward the center of the tube upon centrifugation. Contaminants will move to different positions, and therefore will be separated from the DNA, which can be easily seen and recovered since it will be fluorescent. Subsequent steps will then separate the DNA from the CsCl and ethidium bromide. This technique is particularly effective for separating plasmids – which are small DNA strands that bacteria use to share information on antibiotic resistance with each other, and mitochondrial DNA, and other specific DNA molecules that bind to characteristic amounts of ethidium bromide, and therefore will have unique densities. It is easy to separate mitochondrial and plasmid DNA. These nucleic acids are arranged in highly coiled (called supercoiled) circular patterns. DNA that is not supercoiled (like much longer chromosomal DNA species) bind to different amounts of ethidium bromide, and have a different density – making them easy to separate from the plasmid DNA.
2. Guanidinium Thiocyanate-Phenol-Chloroform nucleic acid extraction
An aqueous solution of the salt guanidinium thiocyanate, when mixed with solvents phenol and chloroform, allows for effective purification of RNA through a minimal number of steps. When samples are ground up and mixed with chloroform, phenol, sodium acetate and guanidinium thiocyanate, and subjected to centrifugation, the salt solution will separate from the solvents – providing an upper aqueous phase and a lower organic phase that consists of the solvents. RNA will remain in the aqueous phase, while proteins, other nucleic acids, and other contaminants will move to the organic phase or to the interface between the two phases.
3. Cetyltrimethylammonium Bromide nucleic acid extraction
This technique is mostly used for plant samples as well as their parts like grains, seeds and leaves. It is also used for many food samples. Nucleic acids, and some other polysaccharides are insoluble in solutions of 2% Cetyltrimethylammonium Bromide nucleic acid extraction (CTAB), at high pH. Neutral polysaccharides and proteins are soluble. This provides an easy way to remove many difficult plant-based contaminants prior to further purification.
4. Chelex® Extraction
This technique is utilized in the field of forensics for DNA extraction from different sources like buccal swabs, blood stain cards and hair. This method uses a resin (trade named Chelex®, that binds to common inhibitors of the polymerase chain reaction (PCR) process. This yields a fairly crude sample, but one that preserves DNA and renders it useful for PCR-based forensic analysis.
5. Alkaline Extraction
This technique is used for plasmid DNA isolation, by itself for relatively crude preparations, or as the first step in virtually all plasmid purification processes. It involves harvesting bacteria of interest from a culture media and consequently exposing the bacteria to a highly alkaline solution. This is generally followed by mixing the alkaline extract with the detergent Sodium Dodecyl Sulfate, which removes proteins and most other contaminants. One of the biggest advantages of this methods is that, since there are many proteins bound to the large chromosomal DNA, this DNA is removed along with the proteins. Since molecular cloning techniques depend on manipulating plasmid DNA, removing the chromosomal DNA is critical.
Solid-phase nucleic acid extraction
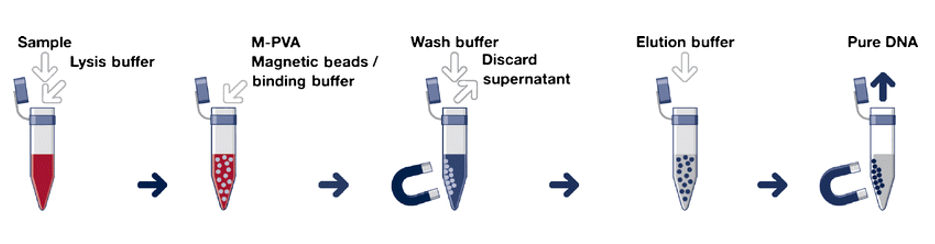
Solid phase extraction methods work by causing nucleic acids to bind to solid supports, such as magnetic beads coated with silica or other materials. The beads (or other supports) are then washed with alcohol to remove contaminants. The support is then washed with a liquid that renders the nucleic acids soluble again, which frees the DNA from the support in a process known as elution. A simple method is to add a solution containing a chaotrope to the crude nucleic acid extract. Chaotropic agents are molecules that disrupt hydrogen bonding network between water molecules. In the presence of chaotropes, nucleic acids before far less soluble and will bind to glass, silica-coated magnetic beads and other solid supports – making it easy to separate them from contaminants. Solid phase extraction methods can also depend on selective binding of DNA and RNA to ion-exchange resins or other chemistries.
Devices
There are numerous devices that are used in nucleic acid extraction methods, these are Spin Columns, Beads or Magnetic Beads, Automated Nucleic Acid Extraction Systems or Liquid Handling Robots as well as Lab-on-a-Chip Cartridges and Microfluidics.
Conclusion
Nucleic acid extraction is an important part of molecular biology. There have been several modifications to the original method which was developed in 1869. Both the mechanical and chemical processes have peculiarities that affect their use, particularly point-of-care diagnostics.
출처 : https://www.biochain.com/general/nucleic-acid-extraction-methods/
'형광면역분석_PCR' 카테고리의 다른 글
| DNA & RNA (0) | 2022.06.22 |
|---|---|
| Fluorescence Fundamentals & PCR Gene Channels (0) | 2022.06.22 |
| PCR(Polymerase chain reaction) 중합효소 연쇄반응 (0) | 2022.05.24 |
| 형광면역분석(법)/fluorescence immunoassay (1) | 2020.11.16 |
| PCR 광학계 구성 및 Gene-Channels (1) | 2020.11.03 |



